| 会议详情 |
|

表观遗传学 (Epigenetics) 是论及决定细胞时代间基因组转录记忆的与 DNA 序列改变无关的信号,介导者和机制的学科。表观遗传事件通过对 DNA 和组蛋白的共价修饰,以转录的 RNA 和翻译的蛋白的方式,调节和功能化地展示编码在 DNA 序列中遗传的结构信息,继而产生在时空多维空间无限多样的表型。健康表观遗传稳态是确保高等生物发育过程中细胞分化和谱系化正常开展所依据的优态基因组表达的必要前提。基因组表观遗传信息界面的异常是包括肿瘤,代谢类和神经系统疾病在内的疾患的病因, 病理学机制之一。
第四届发育和疾病的表观遗传学上海国际研讨会将于 2018 年 9 月 18-21 日,在上海举行。为了加大学术交流的力度,除了 6 个主题 /36 个 25 分钟报告以外,我们还安排了 12 个 10 分钟报告。后者是从两个时长 1.5 小时的墙报摘要者中遴选出来的。此次,与会者将受邀于 19 日和 20 日晚与演讲嘉宾共进晚宴,获得优秀墙报证书和 500 元的现金奖励。
时间地点:
时间: 2018-09-18 至 2018-09-21
地点:复旦大学附属中山医院 18 号楼 3 楼福庆厅(上海市徐汇区枫林路 179 号)
大会主题:
1. DNA 修饰的化学和生物学主题;
2. 组蛋白修饰的化学和生物学主题;
3. 转录的化学生物学和表观调控主题
4. 临床表观遗传学主题;
5. 染色质高级结构的化学和生物学主题;
6. 发育的表观遗传学
 生物360
生物360
生物360,生物医药产业媒体平台,提供生物医药领域最新媒体信息,精品专题策划,人才对接互动等综合服务。中文生命科学界资讯站,是中国领先的即时生命科学资讯站点,已成为重要的生物医药信息集散地,提供生命科学业界资讯、评论、观点、产品和策划。 我们的核心竞争力:快速响应;报道立场公正中立;尽可能提供关联信息;网友讨论气氛浓厚。 我们致力于奉献适合中文读者阅读的新闻资讯,呈现生命科学如何影响人们生活的报道。
18th, Sept, 2018 13:00-17:30 会前会 | ||
DNA 甲基化专题研讨会 | ||
| 13:00-14:00 | 会议注册 | |
| 14:00-14:30 | DNA 甲基化在皮肤病研究中的进(TBD) | 张学军教授安徽医科大学 |
| 14:30-15:00 | DNA 甲基化在液体活检中的应用 | 戴珩 博士中科普瑞 CTO |
| 15:00-15:30 | 甲基化在鼻咽癌中的预后作用及机制研究 | 柳娜 博士(马骏教授团队) 中山大学肿瘤防治中心 |
| 15:30-15:50 | 茶歇 | |
| 15:50-16:20 | Epigenome-wide association study identifies Behcet's diseaseassociated methylation loci in Han Chinese | 于红松 教授遵义医学院 |
| 16:20-17:00 | 后基因组时代下的甲基化多组学研究策略 | 李明辉 博士 表观星图计划首席科学家 中科普瑞科研服务技术产品总监 |
| 17:00-17:30 | 互动讨论:表观星图项目 | 朱景德、张学军、柳娜、 于红松、戴珩、李明辉 |
19th, Sept, 2018 | ||
| 8:30-8:40 | Opening | |
I. Chemical and Biological Perspectives of DNA Modifications | ||
Chair of the first half part: Kazu Ushijima (Tokyo) | ||
| 8:40-9:05 | A vitamin C-derived DNA modification | Guoliang Xu (Academia Sinica,Shanghai) |
| 9:05-9:25 | Aging, epigenetic clocks and cancer risk | Andrew Teschendorff |
| 9:25-9:50 | Active DNA Demethylation by Vertebrate DNA Methyltransferases | James Shen (AcademiaSinica,Taipei) |
| 9:50-10:00 | The DNA modification landscape of human genome: anexperience of HuaXia1 genome/ Third-Generation Sequencing. | Depeng Wang (GrandomicsBiosciences) |
| 10:00-10:10 | N6-Methyladenine DNA Modification in the Human Genome | Chuanle Xiao (ZhongshanOphthalmic Center, Sun Yat-sen University) |
| 10:10-10:35 | Break+Group Photograph | |
Chair of the second half part : Fei Lan (FuDan University) | ||
| 10:35-11:00 | Structural Insight into dynamic regulation of DNA methylation | Yanhui Xu (FudanUniversity) |
| 11:00-11:25 | DNA 5-Hydroxymethylcytosines from Cell-free Circulating DNAasDiagnostic Biomarkers for Human Cancers | Xingyu Lu (Shanghai) |
| 11:25-11:35 | Bisulfite-free, nano-scale analysis of 5-hydroxymethylcytosine atsingle base resolution. | Hu Zeng(Peking University) |
| 11:35-11:45 | BRIF-seq: bisulfite-converted randomly integrated fragmentssequencing at single cell level | Xiang Li (HuazhongAgricultural University) |
| 11:45-11:55 | The role of differentially methylated regions in normalhematopoiesis and leukemogenesis | Pengxu Qian (ZhejiangUniverisity) |
| 11:55-13:00 | Lunch | |
| 13:00-14:30 | Poster session Chair: Guohong Li | |
| 14:30-14:40 | Decoding the epigenetic landscape by the histone readers:implications in human diseases | Chandrima Das |
| 14:40-14:50 | Epigenome reprogramming as a basis for combination therapy | Naoko Hattori |
II.Chemical and Biological Perspectives of histone modifications | ||
Chair of the first half part : Youngjun Kim(Korea, Seul) | ||
| 14:50-15:15 | Structure and function of yeats domain in health and disease | Haitao Li (TsinghuaUniversity), |
| 15:15-15:40 | The dynamic regulation of histone methylation | Fei Lan (Fudan University) |
| 15:40-16:05 | De-acetylation and Epigenomic regulation | Ullas Kolthur-Seetharam (Tata Institute, India) |
| 16:05-16:30 | Metabolic regulation of gene expression | Zhimin Lu (MD Anderson) |
| 16:30-16:50 | Tea Break | |
Chair of the second half part: Lijung Juan (Academia Sinica, Taipei) | ||
| 16:50-17:15 | Epgenetics and Adipogenesis: Implications of HistonemodificationsLanguages | Tapas K. Kundu (India) |
| 17:15-17:25 | UTX functions as an escape from X-inactivation tumor-suppressorin B cell lymphoma | Hai Jiang (Academica Sinica,Shanghai) |
| 17:25-17:35 | Discovery and Functional Insight of a SIRT6 Allosteric | Xiuyan Yang (Shanghai JiaoTong University School ofMedicine) |
20th, Sept, 2018 | ||
III. Chemical and epigenetic regulation of transcription | ||
Chair of the first half part: Shyam PRABHAKAR (Singapore) | ||
| 8:30-8:55 | R-loopsin physiology and pathology | Xiangdong Fu (UCSD) |
| 8:55-9:20 | Epigenetic Regulation of Hematopoietic Stem Cell Development | Feng Liu (AcademiaSinica,Beijing) |
| 9:20-9:50 | The Diversity and Function of Long Noncoding RNAs | Lingling Chen (AcademiaSinica,Shanghai) |
| 9:50-10:15 | Cis-acting lncRNAs in transcription and chromatin regulation | Xiaohua Shen (TsinghuaUniversity) |
| 10:15-10:40 | Tea Break | |
Chair of the second half part: Hongkui Deng (Peking University) | ||
| 10:40-11:05 | R-loop, the general chromatin feature in Arabidopsis genomes | Qianwen Sun(TsinghuaUniversity) |
| 11:05-11:30 | Re-thinking the coding capacity of non-coding RNA | Zefeng Wang (AcademiaSinica,Shanghai) |
| 11:30-11:55 | A long noncoding RNA in telomere maintenance, gene regulationandinter-chromosomal pairing | Hsueh-Ping Chu (NationalTaiwanUniversity) |
| 11:55-12:05 | Single-cell Landscape during Cell-fate conversion | Yuin-Han Jonathan LOH |
| 12:05-13:10 | Lunch | |
Short-talks Chair: Yanhui Xu | ||
| 13:10-13:20 | A new layer of rRNA regulation by small interference RNAs andthe nuclear RNAi pathway | Shouhong Guang (Universityof Science and Technology ofChina) |
| 13:20-13:30 | m6A methyltransferase complex regulates hematopoietic ste-mcell selfrenewal in the bone marrow | Bo Zhou(AcademiaSinica,Shanghai) |
| 13:30-13:40 | RNA m6A Modifications in T Cell Differentiation and Inflammation | Huabing Li (Shanghai JiaoTong University School ofMedicine) |
| 13:40-13:50 | Function and mechanism of 2OG-oxygenase JMJD6 in breastcarcinogenesis | Wen Liu(Xiamen University ) |
IV.Clinical Epigenetics | ||
Chair of the first half part : Tapas Kundu(India) | ||
| 13:50-14:15 | Implication of Long Non-coding RNAs in Cancer Biology | Yutaka Kondo(NagoyaUniversity, Japan) |
| 14:40-15:05 | Global transcriptional memory is mediated by H3K4me3-Rpd3LHDAC pathway | TaeSoo Kim |
| 15:05-15:30 | Whole-genome and epigenomic landscapes of etiologicallydistinctsubtypes of cholangiocarcinoma | Teh Bin Tean (CancerCenter,Singapore), |
| 15:30-15:55 | Functional prediction of causal regulatory variants identifies anovelautism gene | Jung Kyoon Choi (Korea) |
| 15:55-16:20 | Tea Break | |
Chair of the second half part: Zefeng Wang (Academia Sinica) | ||
| 16:20-16:45 | Histone acetylome-wide association study identifies epigenetic changesand a protective host response mechanism to infection | ShyamPRABHAKAR |
| 16:45-16:55 | Characterization of interaction modes between the BRMbromodomain and inhibitors in the limit of NMR intermediateexchange | Ke Ruan(University of Scienceand Technology of China) |
| 16:55-17:05 | Biochemical studies and molecular dynamic simulations reveal themolecular basis of conformational changes in dnamethyltransferase-1 (dnmt1) | Shijie Chen (Academia Sinica,Shanghai) |
| 17:05-17:15 | Drug discovery of protein-protein interactions by targeting epi-modification sites of untargetable protein | Wenchao Lu ( Academia Sinica,Shanghai) |
21st, Sept, 2018 | ||
V.Chemical and biological perspectives of the higher-orderchromatin structure | ||
Chair of the first half part: Young-Joon Kim(Korea) | ||
| 8:30-8:55 | Crosstalk between histone modifications duringheterochromatinassembly | Jun-ichi Nakayama (Tokyo,Japan) |
| 8:55-9:20 | Dynamic regulation of higher-order chromatin structures ingeneregulation | Guohong Li (AdademiaSinica,Beijing) |
| 9:20-9:45 | Reprogramming of chromatin architecture in earlymammaliandevelopment | Wei Xie (Tsinghua University) |
| 9:45-10:10 | Combinatorial mechanisms in 3D genome folding andtranscriptionregulation | Yijun Ruan (Jackson Lab,USA) |
| 10:10-10:30 | Tea Break | |
Chair of the second half part: Lingling Chen (Academia Sinica,Shanghai) | ||
| 10:30-10:55 | Systematic Mapping of RNA-Chromatin Interactions In Vivo | Sheng Zhong (UCSD) |
| 10:55-11:05 | Multisite substrate recognition in Asf1-dependent acetylation ofhistone H3 K56 by Rtt109 | Lin Zhang ( Academia Sinica,Beijing) |
| 11:05-11:15 | 3D Genome of Multiple Myeloma Reveals Spatial GenomeDisorganization Associated with Copy Number Variations | Cheng Li (Peking University) |
| 11:15-11:25 | Chromatin remodelling during brain development | Weijun Feng (FudanUniversity) |
| 11:25-11:35 | ||
| 11:35-11:45 | A novel RNA epigenetic mechanism: m6A RNA-mediated celldamage response | Chih-Hung Hsu (ZhejiangUniverisity) |
| 11:45-13:00 | Lunch | |
VI. Developmental Epigenetics | ||
Chair of the first half part: Guoliang Xu (Academia Sinica,Shanghai) | ||
| 13:00-13:25 | Epigenetic programming by maternal behavior | Moshe Szyf (Canada, McGillUniversity ) |
| 13:25-13:50 | Single-cell multi-omics sequencing of mouse early embryosandembryonic stem cells | Fuchou Tang (PekingUniversity) |
| 13:50-14:00 | Chromatin Accessibility Landscape in Human Early Embryos andIts Association with Evolution | Lei Gao (Academia Sinica,Beijing) |
| 14:00-14:10 | Dnmt2 mediates intergenerational transmission of paternallyacquired metabolic disorders through sperm small non-codingRNAs | Yunfang Zhang (The ThirdMilitary Medical University) |
| 14:10-14:20 | lncRNA-MIR100HG derived miR-100 and miR-125b mediatecetuximab resistance via Wnt/β-catenin signaling in colorectalcancer | Yuanyuan Lu (The FourthMilitary Medical University) |
| 14:20-14:30 | Stella safeguards the oocyte methylome by preventing excessivede novo methylation mediated by DNMT1 | Zhuqiang Zhang (AcademiaSinica) |
| 14:30-14:50 | Tea Break | |
Chair of the second half part: Xiangdong Fu(UCSD) | ||
| 14:50-15:00 | Spatio-temporal dynamics of chromatin in developmentalhematopoietic cells initiating the oncogenic alterations | Dengli Hong (Shanghai JiaoTong University School ofMedicine) |
| 15:00-15:25 | MAnorm2: quantitative comparison of ChIP-seq samplesongroup level to identify sex-biased histone modification sites | Zhen Shao(AcademiaSinica,Shanghai) |
| 15:25-15:50 | Small Molecule-induced Cell Fate Reprogramming | Hongkui Deng(PekingUniversity) |
| 15:50-16:00 | Closing | |
多人参会优惠: 2 人以上(含 2 人),打 9 折; 4 人以上(含 4 人),打 8.25 折; 7 人以上(含 7 人),打 7.5 折;
| 票务类型 | 07-01 至 09-21 | 当前价格 |
| 学术界 | 2860.00 | 2860.00 |
| 学生 | 1950.00 | 1950.00 |
| 企业 | 3250.00 | 3250.00 |
关于注册费用
关于发票
关于退款
会馆场馆
地址:复旦大学附属中山医院 18 号楼 3 楼福庆厅(上海市徐汇区枫林路 179 号)
酒店住宿:
协议酒店:上海好望角大酒店
地址: 上海徐汇区肇嘉浜路 500 号
酒店电话: 400-172-1188
|
房型 |
市场挂牌价 (RMB/Day) |
参会者优惠价 (RMB/Day) |
|
标准间 |
798 |
请联系询问 |


会场交通路线
火车站 / 机场至酒店
1. 距离上海虹桥火车站约 15.3 公里 乘坐地铁 2 号线广兰路方向 ,在静安寺站下车,换乘地铁 7 号线 ( 花木路方向
),在肇嘉浜路站 下车 (3 号口出),步行约 350 米。乘坐出租车约 53 元,50 分钟。
2. 距离上海火车站 18 公里,乘坐地铁 1 号线(莘庄方向)常熟路 站 下车换乘地铁 7 号线(花木路方向)在肇嘉浜路 站 下车 (3 号口出),步行约 350 米。
3. 距离浦东国际机场 55 公里,乘坐地铁 2 号线东延伸段(广兰路方向)浦东国际机场站上车广兰路站 下车 ,换乘 地铁 2 号线 (徐泾东方向)世纪大道 站 下车 换乘 地铁 9 号线 (松江南站方向), 肇嘉浜路 站 (4 口出) 下车步行约 400 米。距离徐家汇商圈 1 公里,乘坐出租车约 5 -10 分钟。
酒店至会场
从酒店门口右转(向西),沿肇嘉浜路走约 60 米,上天桥后左转,下天桥后沿枫林路直行(向南),约 350 米后到达中山医院 18 号楼。
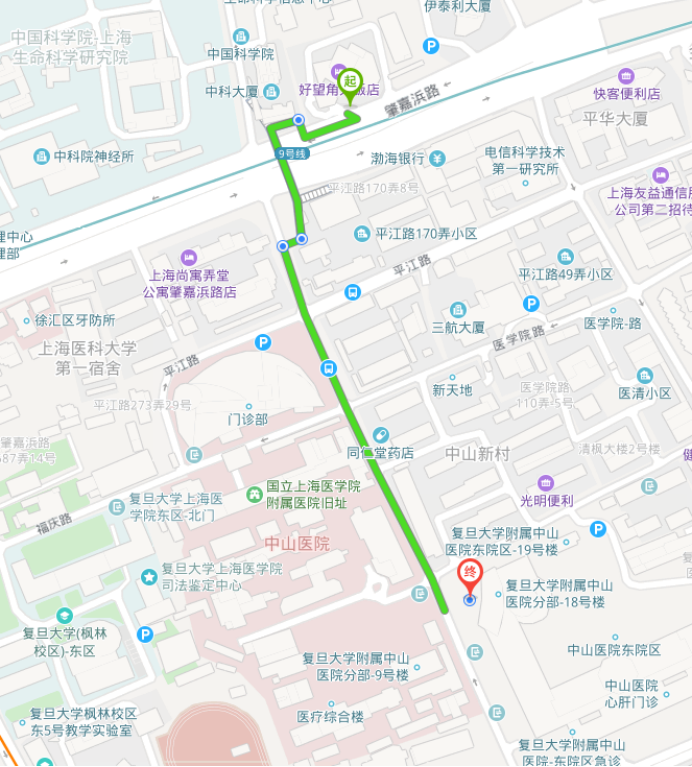
 复旦大学附属中山医院
复旦大学附属中山医院
复旦大学附属中山医院位于上海市枫林路180号/斜土路1609号/松江区佘山镇刘家山村456号/医学院路111号,占地面积95892.1平方米,总建筑面积338732平方米,始建于1937年,是一所三级综合医院,是上海市医保定点单位。
2018年12月4日,被国家卫健委公布为首批肿瘤多学科诊疗试点医院。
相关会议
2026-04-24成都
2026-03-20汕头